Capítulo 4 Acessando e criando Funções, Importação e exportação dados
4.1 Funções
Assista este conteúdo em Cap 3 - Funções no PVANet
Embora existam uma grande variedade de funções e pacotes disponíveis e crescendo em número, a grande versatilidade do R nos permite criar comandos/funções personalizadas.
No entanto, uma dúvida que por ventura você tenha e não consiga sanar dentro das opções de ajuda do próprio R, buscar em fóruns sobre o assunto pode ser uma saída, pois geralmente a sua dúvida foi a dúvida de alguém antes de você.
Vamos inicialmente dar uma olhada nas funções do próprio R.
Até aqui vimos algumas funções como: c(), seq(), rep(), print(), is.numeric(), is.integer(), is.double(), is.character(), typeof(), which(), grep() e etc
Para entender melhor como uma função funciona ou o que ela faz e permite fazer podemos pedir ajuda escrevendo ?nomedafunção.
Por exemplo:
?rnorm #Gera um vetor de números aleatórios normalmente ditribuídos## starting httpd help server ... doneNo display de ajuda da função rnorm()observamos como a função deve ser utilizada, quais parâmetros devem ser inseridos.
rnorm(n, mean = 0, sd = 1)
rnorm(5 ,10 , 8) #Vejam que não estão próximas de zero, mas de dez. ## [1] 22.101881 27.692473 18.294883 8.961472 4.598034Podemos dar nomes aos parâmetros também, caso não sejam nomeados, os parâmetros seguirão a ordem estabelecida na função original inciada em n seguida de mean e sd.
set.seed(1234)
rnorm(n=5 ,mean=10 , sd=8) ## [1] 0.343474 12.219434 18.675529 -8.765582 13.432998set.seed(1234)
rnorm(mean=10,n=5, sd=8) # Uma vez nomeados, eles podem ser arranjados da maneira que desejar. ## [1] 0.343474 12.219434 18.675529 -8.765582 13.432998Quando você executou ?rnorm, você deve ter percebido que existem outro comandos semelhantes:
dnorm(x, mean = 0, sd = 1, log = FALSE)
pnorm(q, mean = 0, sd = 1, lower.tail = TRUE, log.p = FALSE)
qnorm(p, mean = 0, sd = 1, lower.tail = TRUE, log.p = FALSE)
dnorm retorna o valor da função de densidade de probabilidade para os parâmetros. fornececidos.
dnorm(5, mean = 10, sd = 8)## [1] 0.04102012pnorm retorna a integral de -Inf até q de um pdf ( Probability density function) da distribuição normal onde q é o z-score.
pnorm(5, mean = 10, sd = 8)## [1] 0.2659855qnorm é simplesmente o inverso do cdf ( cumulative distribution function), no qual você pode entender com o inverso de pnorm.
qnorm(.2) # Responde como o z-score do vigèsimo elemento de uma distribuiçãop normal.## [1] -0.8416212Para mais detalhes sobre estas funções clique aqui.
As funções acima e tantas outras estão disponíveis no R desde sua instalação, assim como as funções básicas abaixo.
| função | Descrição |
|---|---|
| abs(x) | Valor absoluto |
| sqrt(x) | Raiz quadrada |
| ceiling(x) | Arredonda para o inteiro acima |
| floor(x) | Arredonda para o inteiro abaixo |
| trunc(x) | Trunca para um inteiro |
| round(x,digits=n) | Arredonda de acordo com número de casas decimais |
| log(x) | Logarítimo natural |
| log10(x) | Logarítmo decimal |
| exp(x) | Exponenciação de base e com expoente x |
| sin(x) | Seno em radianos |
| cos(x) | Cosseno em radianos |
| tan(x) | Tangente em radianos |
## [1] 9.99## [1] 1.772454## [1] 4## [1] 3## [1] 3## [1] 3.14## [1] 1.14473## [1] 0.4971499## [1] 23.14069## [1] 1.224606e-16## [1] -1## [1] -1.224647e-16Pesquise por asin(), acos(), atan().
Embora seja rico de opções, com funções dos mais variados tipos e aplicações às vezes não encontramos aquelas específicas que precisamos para algo específico. Ou, mesmo que esteja desenvolvendo uma metodologia nova com novos approaches ou um novo pacote você precisará impreterivelmente de criar suas próprias funções.
As funções às vezes não são tão simples para uma primeira tentativa de uso ou criação principalmente para aqueles que estão iniciando em R ou qualquer linguagem de programação. Podemos imaginar a função dividida em 3 partes (entrada, processamento e saída) como uma caixa com muito buracos de uma lado ( inputs) e apenas um buraco do outro ( output) e a caixa atua de acordo com nossos comandos. Ou ainda como processamento de frutas para um suco.

Estrutura de uma função
Estrutura de uma função:
* A função recebe um nome que recebe os argumentos e comandos a serem executados. O nome da função será o comando a ser executado sempre que desejado.
Após o atribuidor vem o comando function seguindo de (). Dentro de () vem os argumentos (posição ou nomes) que são as variáveis ou outras informações inseridas pelo usuário.
Após fechar os parênteses abre-se chaves {} onde entram as equações ou testes a serem executados e as opções de saídas com o comando return quer geralmente encontra-se ao final da função. Não há restrição sobre a estrutura ou tipo de objeto retornado (
list, data.frame, matrix, numeric, logical… etc).Após o registro da função no ambiente do R (Veja o global environment), a função fica pronta para uso.
minhafunção <- function(arg1, arg2, ... ){
statements
return(object)
}Vamos criar uma função de divisão.
divisao <- function(x, y) {
resultado <- x/y
print(resultado)
}- A função se chama
divisao. - Declare as variáveis x, y entre ();
- {} guardam os comandos a serem executados;
resultado <- x/yeprint(resultado)são os comandos a serem executados.
##
## Attaching package: 'flow'## The following object is masked _by_ '.GlobalEnv':
##
## ddivisao(50,25)## [1] 2divisao(22,4.4)## [1] 5g<-5
x<-10
a<-x
divisao(g,a)## [1] 0.5divisao(a+g,g)## [1] 3Experimentem e vejam o que acontece.
divisao(50,)## Error in divisao(50, ): argumento "y" ausente, sem padrãodivisao(50,25,32)## Error in divisao(50, 25, 32): unused argument (32)divisao(a,m)## , , 1
##
## [,1] [,2] [,3]
## [1,] 10.000000 2.000000 1.1111111
## [2,] 5.000000 1.666667 1.0000000
## [3,] 3.333333 1.428571 0.9090909
## [4,] 2.500000 1.250000 0.8333333
##
## , , 2
##
## [,1] [,2] [,3]
## [1,] 0.7692308 0.5882353 0.4761905
## [2,] 0.7142857 0.5555556 0.4545455
## [3,] 0.6666667 0.5263158 0.4347826
## [4,] 0.6250000 0.5000000 0.4166667***Escreva uma função teste chamada multiplicador que multiplica os valores 12.8, 19.2 e pi. _pi_é armazenado internamente pelo R com 6 casas decimais e pode ser chamada escrevendo pi.***
multiplicador <- function(x, y, z) {
resultado <- x*y*z
print(resultado) } # `print` mostra o resultado de um objeto ou operação
multiplicador(12.8, 19.2, pi)## [1] 772.0778Escreva uma função teste chamada multiplicador que multiplica os valores um vetor com 50 números por outro de 50 números e armazene o resultado como G sem mostrá-los na tela
s<-seq(from= 1,to=10, length.out=50);
length(s) ## [1] 50s## [1] 1.000000 1.183673 1.367347 1.551020 1.734694 1.918367 2.102041
## [8] 2.285714 2.469388 2.653061 2.836735 3.020408 3.204082 3.387755
## [15] 3.571429 3.755102 3.938776 4.122449 4.306122 4.489796 4.673469
## [22] 4.857143 5.040816 5.224490 5.408163 5.591837 5.775510 5.959184
## [29] 6.142857 6.326531 6.510204 6.693878 6.877551 7.061224 7.244898
## [36] 7.428571 7.612245 7.795918 7.979592 8.163265 8.346939 8.530612
## [43] 8.714286 8.897959 9.081633 9.265306 9.448980 9.632653 9.816327
## [50] 10.000000d<-seq(from=1,to=10,length.out = length(s))
d## [1] 1.000000 1.183673 1.367347 1.551020 1.734694 1.918367 2.102041
## [8] 2.285714 2.469388 2.653061 2.836735 3.020408 3.204082 3.387755
## [15] 3.571429 3.755102 3.938776 4.122449 4.306122 4.489796 4.673469
## [22] 4.857143 5.040816 5.224490 5.408163 5.591837 5.775510 5.959184
## [29] 6.142857 6.326531 6.510204 6.693878 6.877551 7.061224 7.244898
## [36] 7.428571 7.612245 7.795918 7.979592 8.163265 8.346939 8.530612
## [43] 8.714286 8.897959 9.081633 9.265306 9.448980 9.632653 9.816327
## [50] 10.000000multiplicador <- function(x, y){
resultado <- x*y
assign("resultado", resultado, envir=globalenv())
} # `assign` atribui os valores de uma operação a um objeto
multiplicador(s,d)
resultado## [1] 1.000000 1.401083 1.869638 2.405664 3.009163 3.680133
## [7] 4.418576 5.224490 6.097876 7.038734 8.047064 9.122865
## [13] 10.266139 11.476885 12.755102 14.100791 15.513953 16.994586
## [19] 18.542691 20.158267 21.841316 23.591837 25.409829 27.295294
## [25] 29.248230 31.268638 33.356518 35.511870 37.734694 40.024990
## [31] 42.382757 44.807997 47.300708 49.860891 52.488546 55.183673
## [37] 57.946272 60.776343 63.673886 66.638900 69.671387 72.771345
## [43] 75.938776 79.173678 82.476052 85.845898 89.283215 92.788005
## [49] 96.360267 100.000000media <- function( x = c( 1, 1, 1, 1)){
#Neste caso utilizamos valores default que permitem testes caso algo saia errado
#Calcula média do inpout x
out <- sum(x)/length(x)
return( out) # `return` devolve/retorna/mostra os resultado de uma operação ou objeto
}
minha.media <- media( x = 1: 100) # print result print( my.mean)
print(minha.media)## [1] 50.5As funções também permitem maior interação com usuário para a tomada de uma decisão por exemplo.
mensagem <- function( arg1=readline(prompt="escreva sua mensagem: "), arg2 = readline(prompt="De novo: ")){
msg1 <- paste0(arg1)
msg2 <- paste0(arg2)
cat( msg1, msg2)
}
mensagem()## escreva sua mensagem:
## De novo:
## 4.2 Pacotes
Assista este conteúdo em Cap 3 - Pacotes no PVANet
4.2.1 O que são? De onde vem? Para onde vão? Do que se alimentam?
Quando iniciamos o R pouco mais de 2000 funções e outros objetos na memória estão prontos para uso.
No R temos os pacotes que são feitos para cálculos estatísticos dos mais variados, pacotes para fazer gráficos, pacotes aplicados à um determinado tipo de dado ou subdivisão da ciência e etc…
Eles basicamente permitem vocês expandirem a funcionalidade do R para programação, ao invés de escrever dezenas de linhas de códigos, esses códigos são preparados, empacotados e tudo que você precisa fazer e usá-lo.
Os Packages são coleções de funções, dados, e códigos compilados num formato bem definido. O diretório onde os pacotes estão armazenados são chamados library. A função
.libPaths() onde sua biblioteca está alojada.
.libPaths()## [1] "C:/Users/jacks/AppData/Local/R/win-library/4.2"
## [2] "C:/Program Files/R/R-4.2.3/library"Milhares de outras funções, para as mais diversas tarefas, podem ser adicionadas por meio de pacotes (packages) disponíveis livremente na internet.
No entanto, a principal fonte de pacotes para o R é o CRAN, mas também temos r-forge, github e outras fontes pela internet.
CRAN now has 10,000 R packages. Here’s how to find the ones you need.
!!!!!!!!!! Não é porque um pacote está no CRAN que ele foi testado e funciona perfeitamente!!!!!!!!!
Para instalar um novo pacote, utilize a função install.packages("nome do pacote") ou
Tools->Install Packages->Install from (Repository CRAN) -> Packages (escreva o nome do pacote).
Para carregar na memória um pacote já instalado, library(nome do pacote) ou require(nome do pacote).
Ao abrir lista de pacotes vemos quais estão disponíveis. Os que estão marcados estão ativados e disponíveis para uso. Aqueles que não estão marcados podem ser ativados e suas funções utilizadas.
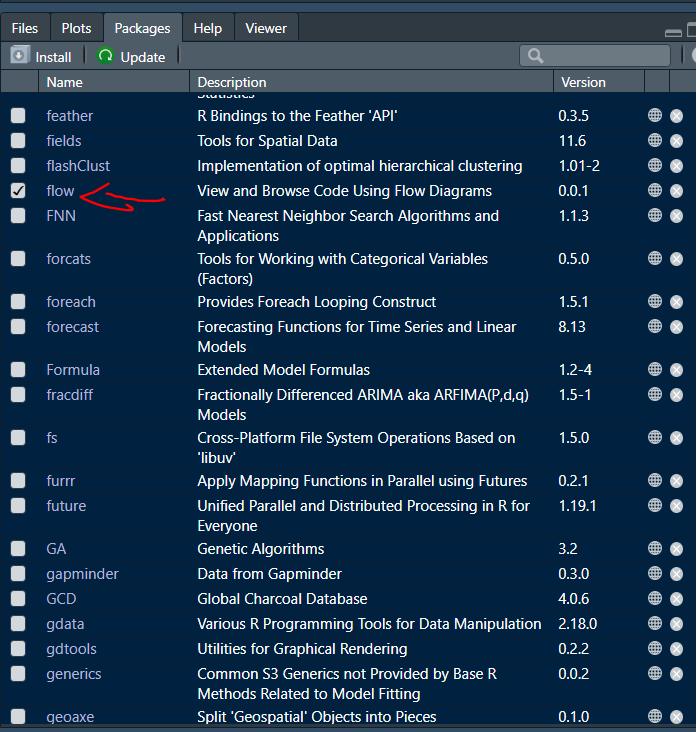
lista de pacotes instalados
Vamos deletar o pacote ggplot2, basta clicar no x no lado direito.
Agora vamos reinstalar pela barra de ferramentas.
Mas o que CRAN quer dizer? É o local onde os pacotes são armazenados.
R é um open source software então se você quiser, pode criar um pacote ar mazená-lo no CRAN.
#install.packages("analogue")
library(analogue)## Carregando pacotes exigidos: vegan## Carregando pacotes exigidos: permute## Carregando pacotes exigidos: lattice## This is vegan 2.6-4## analogue version 0.17-6Visite Analogue e explore o que há disponível para o pacote analogue. Use a aba packages para explorar suas funcionalidades
Muitas vezes os pacotes disponíveis no CRAN não estão UpToDate por isso talvez seja mais interessante isntalar os pacotes diretamente do github.
Veja o pacote packman e vamos instalá-lo pelo github. Mas antes vamos instalar o pacote devtools que , entre outras coisas, nos permite “buscar” os pacotes do github.
#install.packages("devtools")
devtools::install_github("trinker/pacman")## Skipping install of 'pacman' from a github remote, the SHA1 (ace09364) has not changed since last install.
## Use `force = TRUE` to force installationAo utilizar o software R e um pacote na execução de seu trabalho você deve citá-los.
citation () # citação do software R##
## To cite R in publications use:
##
## R Core Team (2023). R: A language and environment for statistical
## computing. R Foundation for Statistical Computing, Vienna, Austria.
## URL https://www.R-project.org/.
##
## A BibTeX entry for LaTeX users is
##
## @Manual{,
## title = {R: A Language and Environment for Statistical Computing},
## author = {{R Core Team}},
## organization = {R Foundation for Statistical Computing},
## address = {Vienna, Austria},
## year = {2023},
## url = {https://www.R-project.org/},
## }
##
## We have invested a lot of time and effort in creating R, please cite it
## when using it for data analysis. See also 'citation("pkgname")' for
## citing R packages.citation(package = "analogue") # citação do pacote Analogue##
## To cite use of 'analogue' in publications use:
##
## Simpson, G.L. and Oksanen, J. ( 2021 ). analogue: Analogue matching
## and Modern Analogue Technique transfer function models. (R package
## version 0.17-6 ). (https://cran.r-project.org/package=analogue).
##
## Simpson, G.L. (2007). Analogue Methods in Palaeoecology: Using the
## analogue Package Journal of Statistical Software, 22(2), 1--29
##
## To see these entries in BibTeX format, use 'print(<citation>,
## bibtex=TRUE)', 'toBibtex(.)', or set
## 'options(citation.bibtex.max=999)'.Aqui listo os pacotes carregados agora em meu R.
(.packages())## [1] "analogue" "vegan" "lattice" "permute" "flow" "purrr"
## [7] "magrittr" "ggplot2" "stats" "graphics" "grDevices" "utils"
## [13] "datasets" "methods" "base"Aqui listo os pacotes instalados em meu R.
meus.pacotes <- library()$results[,1]
meus.pacotes## [1] "abind" "ade4" "AGD"
## [4] "alphavantager" "ambient" "Amelia"
## [7] "AmesHousing" "analogue" "anytime"
## [10] "ape" "arm" "ash"
## [13] "askpass" "aspace" "assertthat"
## [16] "audio" "automap" "babynames"
## [19] "backports" "base64enc" "beepr"
## [22] "bfast" "bfastSpatial" "BH"
## [25] "bigleaf" "bit" "bit64"
## [28] "bitops" "blob" "bookdown"
## [31] "brew" "brglm" "brio"
## [34] "broom" "BSDA" "bslib"
## [37] "cachem" "CALIBERrfimpute" "callr"
## [40] "car" "carData" "caret"
## [43] "caTools" "cellranger" "checkmate"
## [46] "classInt" "cli" "climateStability"
## [49] "climdex.pcic" "climdex.pcic.ncdf" "clipr"
## [52] "clock" "clue" "cluster"
## [55] "clv" "coda" "colorspace"
## [58] "combinat" "commonmark" "conditionz"
## [61] "conflicted" "contfrac" "coro"
## [64] "corrplot" "cowplot" "cpp11"
## [67] "crayon" "credentials" "crosstalk"
## [70] "crsmeta" "crul" "Cubist"
## [73] "curl" "dados" "data.table"
## [76] "datawizard" "DBI" "dbplyr"
## [79] "decido" "deepnet" "deldir"
## [82] "dendextend" "DEoptimR" "desc"
## [85] "deSolve" "devEMF" "devtools"
## [88] "diagram" "dials" "DiceDesign"
## [91] "dichromat" "diffobj" "digest"
## [94] "dismo" "distributional" "doParallel"
## [97] "doRNG" "doSNOW" "dotCall64"
## [100] "downlit" "downloader" "dplyr"
## [103] "DT" "dtplyr" "dtw"
## [106] "DynACof" "e1071" "earth"
## [109] "easyGgplot2" "editData" "elevatr"
## [112] "ellipse" "ellipsis" "elliptic"
## [115] "emmeans" "ENMeval" "ENMTools"
## [118] "estimability" "evaluate" "evtree"
## [121] "exactextractr" "expm" "extraDistr"
## [124] "fable" "fabletools" "factoextra"
## [127] "FactoMineR" "fansi" "farver"
## [130] "fastmap" "fBasics" "feasts"
## [133] "fields" "flashClust" "flextable"
## [136] "flow" "flowr" "FNN"
## [139] "fontawesome" "fontBitstreamVera" "fontLiberation"
## [142] "fontquiver" "forcats" "foreach"
## [145] "forecast" "forecastHybrid" "Formula"
## [148] "fracdiff" "frbs" "fs"
## [151] "fueleconomy" "functional" "furrr"
## [154] "future" "future.apply" "gamlss"
## [157] "gamlss.data" "gamlss.dist" "gapminder"
## [160] "gargle" "gbm" "GCD"
## [163] "gclus" "gdalUtils" "gdtools"
## [166] "generics" "geobr" "geojsonsf"
## [169] "geometries" "geometry" "gert"
## [172] "gfonts" "ggbiplot" "ggforce"
## [175] "ggformula" "ggmap" "ggOceanMaps"
## [178] "ggOceanMapsData" "ggplot2" "ggpubr"
## [181] "ggrepel" "ggridges" "ggsci"
## [184] "ggsignif" "ggspatial" "ggstance"
## [187] "gh" "GISTools" "git2r"
## [190] "gitcreds" "glmnet" "globals"
## [193] "glue" "goftest" "googledrive"
## [196] "googlesheets4" "gower" "GPfit"
## [199] "gridBase" "gridExtra" "gss"
## [202] "gstat" "gtable" "GWmodel"
## [205] "h2o" "hardhat" "haven"
## [208] "here" "hexbin" "highr"
## [211] "HistogramTools" "Hmisc" "hms"
## [214] "htmlTable" "htmltools" "htmlwidgets"
## [217] "hts" "httpcode" "httpuv"
## [220] "httr" "httr2" "hydroGOF"
## [223] "hydroTSM" "hypergeo" "ids"
## [226] "igraph" "imputeMissings" "infer"
## [229] "ini" "insight" "installr"
## [232] "interp" "intervals" "inum"
## [235] "ipred" "isoband" "iterators"
## [238] "itertools" "janitor" "jpeg"
## [241] "jquerylib" "jsonify" "jsonlite"
## [244] "JuliaCall" "kableExtra" "Kendall"
## [247] "kernlab" "kknn" "knitr"
## [250] "KRLS" "labdsv" "labeling"
## [253] "labelled" "laeken" "Lahman"
## [256] "later" "latticeExtra" "lava"
## [259] "lavaan" "lazyeval" "leafem"
## [262] "leaflet" "leaflet.providers" "leafsync"
## [265] "leaps" "LearnBayes" "lhs"
## [268] "libcoin" "lifecycle" "linprog"
## [271] "listenv" "lme4" "lmom"
## [274] "lmomco" "Lmoments" "lmtest"
## [277] "locfit" "locpol" "longitudinalData"
## [280] "lpSolve" "lsr" "lubridate"
## [283] "lwgeom" "magic" "magrittr"
## [286] "mapproj" "maps" "maptools"
## [289] "markdown" "Matrix" "MatrixModels"
## [292] "matrixStats" "maxnet" "memoise"
## [295] "Metrics" "mi" "mice"
## [298] "mime" "miniUI" "minqa"
## [301] "misc3d" "missForest" "mlbench"
## [304] "mnormt" "modeest" "modeldata"
## [307] "modelenv" "ModelMetrics" "modelr"
## [310] "monmlp" "moonBook" "mosaic"
## [313] "mosaicCore" "mosaicData" "multcompView"
## [316] "munsell" "mvoutlier" "mvpart"
## [319] "MVPARTwrap" "mvtnorm" "nabor"
## [322] "nasaweather" "NbClust" "ncdf4"
## [325] "ncdf4.helpers" "NeuralNetTools" "nloptr"
## [328] "nngeo" "nomnoml" "nortest"
## [331] "numDeriv" "nycflights13" "oai"
## [334] "OCNet" "officer" "openssl"
## [337] "openxlsx" "optimx" "Orcs"
## [340] "packrat" "pacman" "padr"
## [343] "paleofire" "palmerpenguins" "palr"
## [346] "parallelly" "params" "parsnip"
## [349] "partykit" "patchwork" "pbivnorm"
## [352] "pbkrtest" "PCICt" "pdc"
## [355] "PerformanceAnalytics" "permute" "pillar"
## [358] "pixarfilms" "pixmap" "pkgbuild"
## [361] "pkgconfig" "pkgdown" "pkgload"
## [364] "plogr" "plotly" "plotmo"
## [367] "plotrix" "plyr" "png"
## [370] "polyclip" "polynom" "praise"
## [373] "prettymapr" "prettyunits" "princurve"
## [376] "pROC" "processx" "prodlim"
## [379] "profileModel" "profvis" "progress"
## [382] "progressr" "PROJ" "proj4"
## [385] "promises" "proxy" "PRROC"
## [388] "ps" "psych" "purrr"
## [391] "qpdf" "quadmesh" "quadprog"
## [394] "Quandl" "quantmod" "quantreg"
## [397] "quarto" "R.cache" "R.methodsS3"
## [400] "R.oo" "R.utils" "R6"
## [403] "radix" "ragg" "randomForest"
## [406] "rangeModelMetadata" "ranger" "rapidjsonr"
## [409] "rappdirs" "raster" "rasterVis"
## [412] "rayimage" "rayrender" "rayshader"
## [415] "rayvertex" "rbibutils" "rbison"
## [418] "rcmdcheck" "RColorBrewer" "Rcpp"
## [421] "RcppArmadillo" "RcppEigen" "RcppProgress"
## [424] "RcppRoll" "RcppThread" "RcppTOML"
## [427] "RCurl" "Rdpack" "readr"
## [430] "readxl" "rebird" "recipes"
## [433] "rematch" "rematch2" "remotes"
## [436] "repr" "reprex" "reproj"
## [439] "reshape" "reshape2" "rgbif"
## [442] "rgdal" "rgeos" "rgl"
## [445] "RgoogleMaps" "rgrass" "rgrass7"
## [448] "ridigbio" "riingo" "rio"
## [451] "rioja" "rivnet" "rJava"
## [454] "rjson" "rkt" "rlang"
## [457] "rmarkdown" "rmutil" "rnaturalearth"
## [460] "rnaturalearthdata" "rnaturalearthhires" "rngtools"
## [463] "robustbase" "rosm" "roxygen2"
## [466] "rpart.plot" "rprojroot" "rrtable"
## [469] "rsample" "rsconnect" "RSQLite"
## [472] "rstatix" "RStoolbox" "rstudioapi"
## [475] "rticles" "Rtsne" "rversions"
## [478] "rvertnet" "rvest" "rvg"
## [481] "rworldmap" "s2" "sandwich"
## [484] "sass" "scales" "scatterplot3d"
## [487] "sdm" "sdmpredictors" "selectr"
## [490] "servr" "sessioninfo" "sf"
## [493] "sfheaders" "sftime" "sgeostat"
## [496] "shape" "shapefiles" "shiny"
## [499] "shinyBS" "shinydashboard" "shinyjs"
## [502] "shinythemes" "shinyWidgets" "sjlabelled"
## [505] "sjmisc" "skimr" "slickR"
## [508] "slider" "slippymath" "smoothr"
## [511] "snakecase" "snow" "solartime"
## [514] "sourcetools" "sp" "spacefillr"
## [517] "spacetime" "spam" "SparseM"
## [520] "spatialreg" "spatstat" "spatstat.core"
## [523] "spatstat.data" "spatstat.explore" "spatstat.geom"
## [526] "spatstat.linnet" "spatstat.model" "spatstat.random"
## [529] "spatstat.sparse" "spatstat.utils" "spData"
## [532] "spdep" "SPEI" "spex"
## [535] "splancs" "spocc" "spThin"
## [538] "SQUAREM" "SSN" "stable"
## [541] "stabledist" "stars" "statip"
## [544] "stringi" "stringr" "strucchangeRcpp"
## [547] "styler" "svglite" "sys"
## [550] "sysfonts" "systemfonts" "tcltk2"
## [553] "TeachingDemos" "tensor" "terra"
## [556] "terrainmeshr" "testthat" "textshaping"
## [559] "thematic" "thief" "tibble"
## [562] "tidymodels" "tidyquant" "tidyr"
## [565] "tidyselect" "tidyverse" "timechange"
## [568] "timeDate" "timeSeries" "timetk"
## [571] "tinytex" "TLMoments" "tmap"
## [574] "tmaptools" "torch" "traudem"
## [577] "trend" "triebeard" "TSclust"
## [580] "TSdist" "tseries" "tsfeatures"
## [583] "tsibble" "tsibbledata" "TSstudio"
## [586] "TTR" "tune" "tweenr"
## [589] "tzdb" "udunits2" "unikn"
## [592] "units" "urca" "urlchecker"
## [595] "urltools" "usdm" "usethis"
## [598] "utf8" "uuid" "vcd"
## [601] "vctrs" "vegan" "VIM"
## [604] "vip" "viridis" "viridisLite"
## [607] "visdat" "vroom" "waldo"
## [610] "warp" "weathermetrics" "webr"
## [613] "webshot" "wellknown" "wesanderson"
## [616] "whisker" "widgetframe" "withr"
## [619] "wk" "woeBinning" "workflows"
## [622] "workflowsets" "xfun" "xgboost"
## [625] "xlsx" "xlsxjars" "XML"
## [628] "xml2" "xopen" "xtable"
## [631] "xts" "yaml" "yardstick"
## [634] "zeallot" "zip" "zoo"
## [637] "ztable" "zyp" "base"
## [640] "boot" "class" "cluster"
## [643] "codetools" "compiler" "datasets"
## [646] "foreign" "graphics" "grDevices"
## [649] "grid" "KernSmooth" "lattice"
## [652] "MASS" "Matrix" "methods"
## [655] "mgcv" "nlme" "nnet"
## [658] "parallel" "rpart" "spatial"
## [661] "splines" "stats" "stats4"
## [664] "survival" "tcltk" "tools"
## [667] "translations" "utils"Ou poderia simplesmente digitar library().
library()4.3 Entrando com dados
Assista este conteúdo em Cap 3 - Entrando com dados partes 1 e 2 no PVANet
Agora que dominamos 100% as estruturas e tratamento de objetos podemos partir para importação de dados das mais variadas fontes.
Até agora nós basicamente trabalhamos com dados “café com leite”. Criamos uma tabelinha aqui, uma matrizinha acolá um vetor e por aí vai.
Sinto muito em informá-los, mas o mundo é bem mais complexo e cruel.
| “O mundo não é um mar de rosas. É um lugar sujo, um lugar cruel, que não quer saber o quanto você é durão. Vai botar você de joelhos e você vai ficar de joelhos para sempre se você deixar. Você, eu, ninguém vai bater tão forte como a vida, mas não se trata de bater forte. Se trata de quanto você aguenta apanhar e seguir em frente, o quanto você é capaz de aguentar e continuar tentando. É assim que se consegue vencer.Agora se você sabe do teu valor, então vá atrás do que você merece, mas tem que estar preparado para apanhar. E nada de apontar dedos, dizer que você não consegue por causa dele ou dela, ou de quem quer que seja. Só covardes fazem isso e você não é covarde, você é melhor que isso.” |
Balboa, Rocky |
Os dados no R podem ser importados por diferentes meios (teclado, text file, outros softwares estatísticos, planilhas e etc).
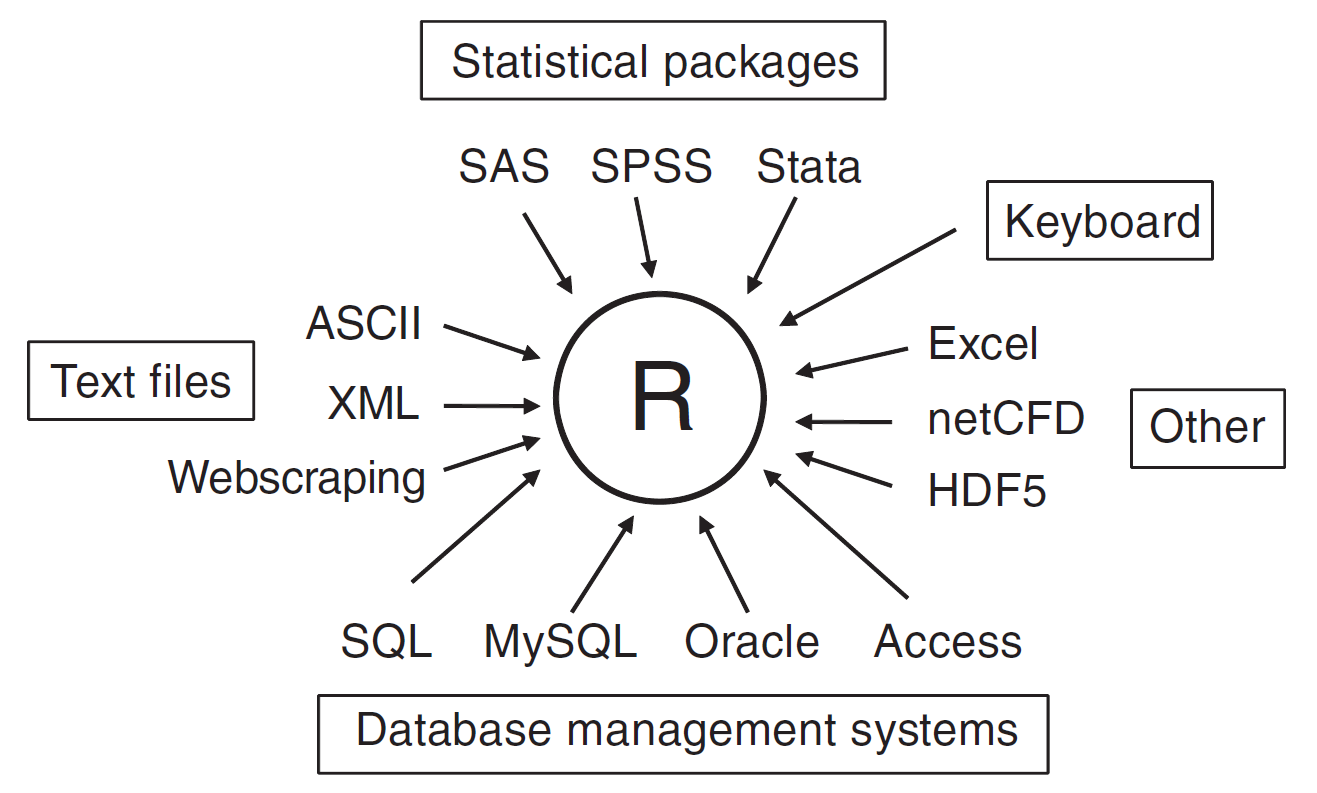
Fonte: Kabacoff (2015)
4.3.1 edit()
A forma mais simples de inserção de dados é através do teclado.
No caso a seguir, o comando Matrícula=numeric(0) cria uma variável de um mode()específico, mas sem dados. O comando edit(), por sua vez, e como nome sugere, permite editar um banco de dados. Um popup abrirá na sua tela que permitirá a sua edição. Após o uso basta fechar que as edições feitas serão salvas.
comando edit()
Alunos.Eng792 <- data.frame(Nome=character(0),Matrícula=numeric(0), Curço=character(0),Nível.de.R=numeric(0),Nível.de.Pós=character(0))
Alunos.Eng792## [1] Nome Matrícula Curço Nível.de.R Nível.de.Pós
## <0 linhas> (ou row.names de comprimento 0)Alunos.Eng792 <- edit(Alunos.Eng792)
Alunos.Eng792## [1] Nome Matrícula Curço Nível.de.R Nível.de.Pós
## <0 linhas> (ou row.names de comprimento 0)4.3.2 built-in
No R também temos os dados buit-in que são dados que já vem “dentro” do R ou pacotes que permite a execução dos exemplos das funções naturais do R e aquelas instaladas por meio dos pacotes. Temos vários dados built-in no R como ìris, mtcars, anscombe, USArrests, USAccDeaths,AirPassengers e etc.
Vamos dar uma olha no USArrests que contém estatísticas de presos por diferentes crimes em EUA em taxas de 100.000.
Presidiarios.USA<-(USArrests)
Presidiarios.USA## Murder Assault UrbanPop Rape
## Alabama 13.2 236 58 21.2
## Alaska 10.0 263 48 44.5
## Arizona 8.1 294 80 31.0
## Arkansas 8.8 190 50 19.5
## California 9.0 276 91 40.6
## Colorado 7.9 204 78 38.7
## Connecticut 3.3 110 77 11.1
## Delaware 5.9 238 72 15.8
## Florida 15.4 335 80 31.9
## Georgia 17.4 211 60 25.8
## Hawaii 5.3 46 83 20.2
## Idaho 2.6 120 54 14.2
## Illinois 10.4 249 83 24.0
## Indiana 7.2 113 65 21.0
## Iowa 2.2 56 57 11.3
## Kansas 6.0 115 66 18.0
## Kentucky 9.7 109 52 16.3
## Louisiana 15.4 249 66 22.2
## Maine 2.1 83 51 7.8
## Maryland 11.3 300 67 27.8
## Massachusetts 4.4 149 85 16.3
## Michigan 12.1 255 74 35.1
## Minnesota 2.7 72 66 14.9
## Mississippi 16.1 259 44 17.1
## Missouri 9.0 178 70 28.2
## Montana 6.0 109 53 16.4
## Nebraska 4.3 102 62 16.5
## Nevada 12.2 252 81 46.0
## New Hampshire 2.1 57 56 9.5
## New Jersey 7.4 159 89 18.8
## New Mexico 11.4 285 70 32.1
## New York 11.1 254 86 26.1
## North Carolina 13.0 337 45 16.1
## North Dakota 0.8 45 44 7.3
## Ohio 7.3 120 75 21.4
## Oklahoma 6.6 151 68 20.0
## Oregon 4.9 159 67 29.3
## Pennsylvania 6.3 106 72 14.9
## Rhode Island 3.4 174 87 8.3
## South Carolina 14.4 279 48 22.5
## South Dakota 3.8 86 45 12.8
## Tennessee 13.2 188 59 26.9
## Texas 12.7 201 80 25.5
## Utah 3.2 120 80 22.9
## Vermont 2.2 48 32 11.2
## Virginia 8.5 156 63 20.7
## Washington 4.0 145 73 26.2
## West Virginia 5.7 81 39 9.3
## Wisconsin 2.6 53 66 10.8
## Wyoming 6.8 161 60 15.64.3.3 scan()
Uma outra forma de entrada manual se dá através dom comando scan(). Neste formato de entrada de dados podemos digitar informação por informação ou copiar de uma fonte e colá-la. Tudo isso feito no console e em formato numérico.
Exemplo com digitação.
teste1 <-scan()
# Digite 1, 2, 4, 5, 6, 7, 8
teste1Exemplo com copia e cola.
seq(30,38)
teste2 <-scan()
teste24.3.4 Lendo arquivos de texto (.txt)
No dia a dia esbarramos em dados que estão disponíveis em formato de texto. Geralmente este dados estão organizados em linhas e colunas onde cada linha representa uma observação e cada coluna uma variável.
Para fazer leitura de dados em formato de texto (.txt) podemos utilizar o comando read.table() que lê o arquivo em formato de tabela e armazena em formato de data.frame.
A sintaxe do comando é:
meu.df <- read.table(file, options)
Existem muitas opções ( options) disponíveis, mas podemos destacar algumas mais utilizadas header, sep, row.names, col.names, skip.
Para saber quais são as options e o que cada uma delas faz você pode acessar através de ?data.datble.
No caso abaixo estamos utilizando sep=",", header=T e skip=3.
Baixe os dados aqui Cap_3_Notas_ENG792.txt no PVANet
notas<-read.table("J:/ENG 792/ENG_792-AVDR/ENG.792-AVDR/Cap_3_Notas_ENG792.txt", sep=",", header = T, skip=3)
notas## Nome Nota.1 Nota.2 Nota.3 Nota.4
## 1 A 10.9 60.9 53.6 12.0
## 2 B 71.3 9.9 13.9 47.0
## 3 C 65.9 0.3 28.9 55.0
## 4 D 3.6 70.0 37.9 75.3
## 5 E 70.9 81.9 43.9 8.6
## 6 F 76.9 53.0 29.9 38.6
## 7 G 73.6 2.3 23.0 37.3
## 8 H 2.0 88.0 10.3 28.3
## 9 I 25.3 11.9 12.3 26.3
## 10 J 7.0 19.3 82.9 46.3
## 11 K 52.0 41.0 70.3 31.6
## 12 L 27.3 74.0 47.6 18.9
## 13 M 87.9 75.9 17.9 80.6
## 14 N 76.0 47.9 2.0 19.6
## 15 O 55.0 60.6 51.9 24.0
## 16 P 76.6 47.9 90.3 55.6
## 17 Q 10.3 55.9 18.0 86.3
## 18 R 17.0 0.9 43.3 14.6
## 19 S 38.6 22.6 23.6 6.6
## 20 T 69.0 31.9 75.3 90.6
## 21 U 26.9 3.6 65.3 48.6
## 22 X 56.6 32.0 74.6 28.0
## 23 Y 22.3 42.3 88.9 65.0
## 24 Z 36.6 44.3 17.9 23.9str(notas)## 'data.frame': 24 obs. of 5 variables:
## $ Nome : chr "A" "B" "C" "D" ...
## $ Nota.1: num 10.9 71.3 65.9 3.6 70.9 76.9 73.6 2 25.3 7 ...
## $ Nota.2: num 60.9 9.9 0.3 70 81.9 53 2.3 88 11.9 19.3 ...
## $ Nota.3: num 53.6 13.9 28.9 37.9 43.9 29.9 23 10.3 12.3 82.9 ...
## $ Nota.4: num 12 47 55 75.3 8.6 38.6 37.3 28.3 26.3 46.3 ...Acima nós criamos o objeto notaspela importação do arquivo Notas_ENG792.txt referente às notas hipotéticas de vocês na disciplina Eng 792 ao término do semestre.
Este arquivo está armazenado em meu computador na pasta J:/ENG 792/ENG_792-AVDR/ENG.792-AVDR/. Preste atenção na forma como ele foi importado o endereço veio antes do nome do arquivo e as separações entre os diretórios se pelo uso de /, mas também poderia ter utilizado \\.
Após o endereço do arquivo vieram as options separadas por vírgulas (,). Se você tiver a curiosidade de abrir o arquivo .txt, você verá como os dados estão organizados e entenderá a utilização de cada argumento.
No entanto, como eu sou gente boa, vou ajudar vocês embora vocês não mereçam.
O argumento sep=","foi utilizado pois os dados de cada “célula” está separado da outra por meio de uma vírgula ,.
sep="" indica espaço delimitando os dados
sep="\t" indica tab delimitando os dados
sep="\n" indica new line delimitaNdo os dados
O argumento header=T" indica que nossa tabela tem cabeçalho e é a primeira linha após o skip=3.
O argumento skip=3" indica que as 3 primeiras linhas de nosso arquivo devem ser ignoradas começando assim, nossa tabela a partir da 4ª linha.
Note ainda que os nomes das colunas foram automaticamente alterados para atender as convenções de condificação do R. Onde originalmente estava Nota 1 agora temos Nota.1.
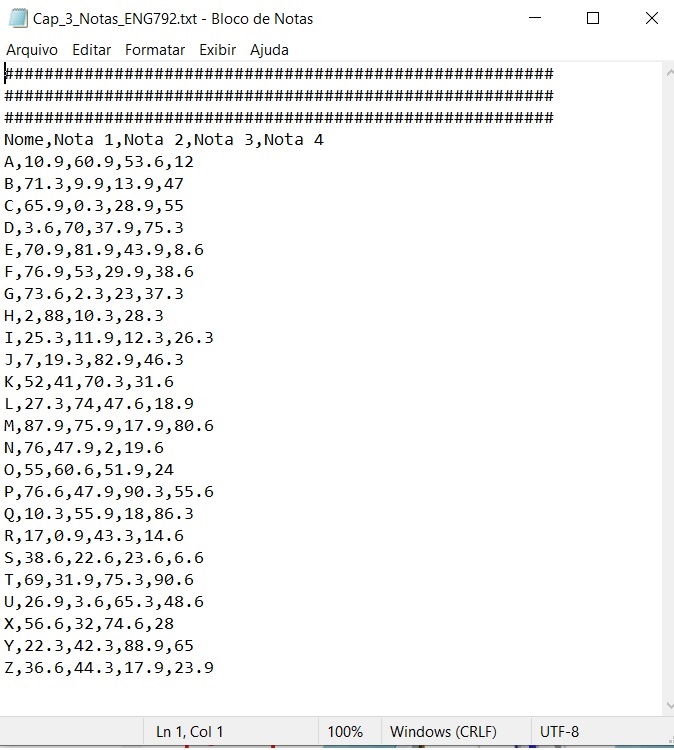
Notas Eng792
RESOLVA ISSO! E digo mais determine que a primeira coluna (os nomes de vocês) sejam o nome das linhas
4.3.5 read.csv()
Embora muito comuns arquivos em formato .txt, a maioria dos cientistas preferem armazenar dados em planilhas eletrônicas como do excel xlsx. ou .csv ( comma-separated values).
Baixe o arquivo Cap_1_P2-Mispriced-Diamonds.csv PVANet
diamantes<-read.csv("J:/ENG 792/ENG_792-AVDR/ENG.792-AVDR/Cap_1_P2-Mispriced-Diamonds.csv")
str(diamantes)## 'data.frame': 53940 obs. of 3 variables:
## $ carat : num 0.23 0.21 0.23 0.29 0.31 0.24 0.24 0.26 0.22 0.23 ...
## $ clarity: chr "SI2" "SI1" "VS1" "VS2" ...
## $ price : int 326 326 327 334 335 336 336 337 337 338 ...head(diamantes) # mostra as 6 primeiras linhas do objeto## carat clarity price
## 1 0.23 SI2 326
## 2 0.21 SI1 326
## 3 0.23 VS1 327
## 4 0.29 VS2 334
## 5 0.31 SI2 335
## 6 0.24 VVS2 336tail(diamantes) # mostra as 6 últimas linhas do objeto ## carat clarity price
## 53935 0.72 SI1 2757
## 53936 0.72 SI1 2757
## 53937 0.72 SI1 2757
## 53938 0.70 SI1 2757
## 53939 0.86 SI2 2757
## 53940 0.75 SI2 2757Observem que no comando read.csv() nós não adicionamos o argumento header=T, issto por que por padrão headeré igual a TRUE (T), caso seus dados não tenham cabeçalhos você deve argumentar header=F
Você já deve ter esbarrado com dados no “brasileiros” nos quais os separadores decimais não são pontos como no padrão internacional, mas vírgulas. Da mesma maneira, em Banânia, os separadores de de células são ponto e vírgula (semi-vírgula - ;).
Para evitar problemas como esse na importação dos dados, (Perlin (2018)) sugere que sejam abertos em um editor de texto qualquer e substitua todos os valores de vírgula (,) por ponto (.). Isso deixa o arquivo em um formato internacional. No Windows, programas como o Notepad e o Notepad + + realizam essa tarefa facilmente, no linux o gedit e etc.
Perlin (2018) suegre para evitar problemas, antes de prosseguir para a importação de dados em um arquivo .csv, deve-se abrir o arquivo em um editor de texto qualquer e verificar:
- A existência de texto antes dos dados e a necessidade de ignorar algumas linhas iniciais;
- A existência ou não dos nomes das colunas;
- O símbolo separador de colunas;
- O símbolo de decimal, o qual deve ser o ponto (.).
Caso não seja possível converter os dados para o formato internacional, para esta jaboticaba você pode utilizar funções específicas como read.csv2() que assume que o indicador de decimal é a vírgula (,) e o separação de colunas é o ponto-e-vírgula (;).
O read.csv()usa como default o padrão internacional sep=”,” e dec=”.” já o read.csv2() tem por default o padrão sep=";" e dec=",".
dec="" (separador de casas decimais)
Experimente ambos comandos para abrir o Cap_3_Notas_ENG792.csv.
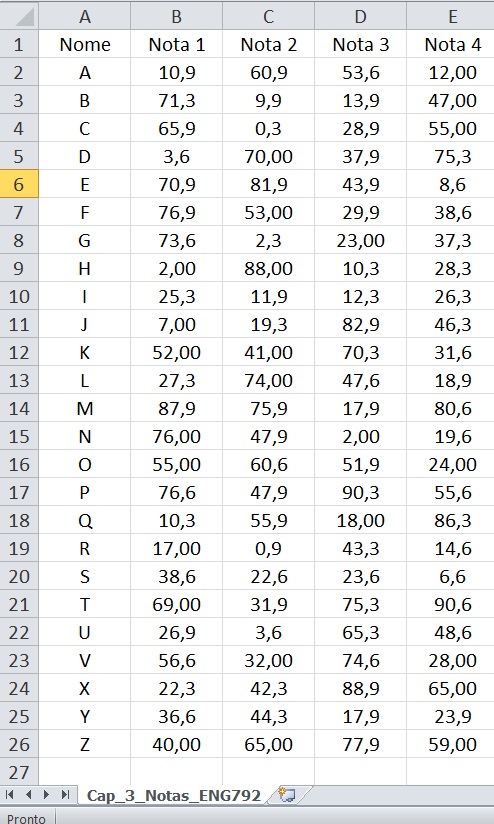
Temos 1 arquivo Cap_3_Notas_ENG792.csv PVANet
Notas.csv<-read.csv2("J:/ENG 792/ENG_792-AVDR/ENG.792-AVDR/Cap_3_Notas_ENG792.csv", row.names = "Nome")
Notas.csv## Nota.1 Nota.2 Nota.3 Nota.4
## A 10.9 60.9 53.6 12.0
## B 71.3 9.9 13.9 47.0
## C 65.9 0.3 28.9 55.0
## D 3.6 70.0 37.9 75.3
## E 70.9 81.9 43.9 8.6
## F 76.9 53.0 29.9 38.6
## G 73.6 2.3 23.0 37.3
## H 2.0 88.0 10.3 28.3
## I 25.3 11.9 12.3 26.3
## J 7.0 19.3 82.9 46.3
## K 52.0 41.0 70.3 31.6
## L 27.3 74.0 47.6 18.9
## M 87.9 75.9 17.9 80.6
## N 76.0 47.9 2.0 19.6
## O 55.0 60.6 51.9 24.0
## P 76.6 47.9 90.3 55.6
## Q 10.3 55.9 18.0 86.3
## R 17.0 0.9 43.3 14.6
## S 38.6 22.6 23.6 6.6
## T 69.0 31.9 75.3 90.6
## U 26.9 3.6 65.3 48.6
## V 56.6 32.0 74.6 28.0
## X 22.3 42.3 88.9 65.0
## Y 36.6 44.3 17.9 23.9
## Z 40.0 65.0 77.9 59.0Notas.csv<-read.csv("J:/ENG 792/ENG_792-AVDR/ENG.792-AVDR/Cap_3_Notas_ENG792.csv", row.names = "Nome")## Error in read.table(file = file, header = header, sep = sep, quote = quote, : more columns than column names
Note que o comando read.csv() não funcionou, faça os ajustes necessários para que ele funcine.
Explore o comandos rowSums(), colSums(), rowMeans(), colMeans().
4.3.6 Planilhas excel completas (.xls ou .xlsx)
É muito comum encontrarmos dados organizados em diferentes planilhas dentro de um mesmo arquivo como no caso do excel ( .xls ou .xlsx) e semelehantes ( .odt).
Estes arquivos e suas diferentes abas podem ser acessados pelo R também, no entanto não há funções nativas que executem esta ação. Precisamos nesse caso instalar pacotes de funções específicas para desempnhar esta operação.
Temos algumas opções de pacotes para esta função como XLConnect (Mirai Solutions GmbH (2020)), xlsx (Dragulescu and Arendt (2020)) e readxl (Hadley Wickham and Bryan (2019)).
Caso a instalação desses pacotes não funcione e, a mensagem de abaixo apareça.
error: No CurrentVersion entry in Software/JavaSoft registry! Try re-installing Java and make sure R and Java have matching architectures.
Experimente instalar ou atualizar antes o Java correspondente ao seu sistema operacional e bit.
Vamos abrir o Definições iniciais - ENG 792.xlsx.
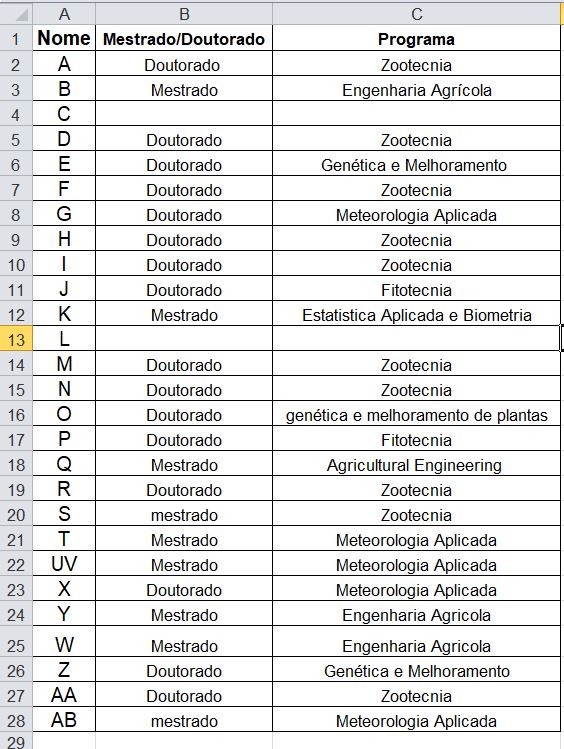
Eu já tenho instalado no meu R, então instale no seu caso não o tenha.
install.packages("xlsx")
Baixe os dados aqui Baixe o arquivo Cap_3_Informações - Alunos ENG 792.xlsx PVANet
library(xlsx)
Info.Alunos<-read.xlsx(file="J:/ENG 792/ENG_792-AVDR/ENG.792-AVDR/Cap_3_Informações - Alunos ENG 792.xlsx", endRow=28,sheetIndex=4,startRow = 2, header = T,encoding="UTF-8")
Info.Alunos4.3.7 Exportar arquivos()
Uma vez que somo capazes de importar arquivos de formatos específicos, somo também capazes de exportá-los.
Podemos exportar data.frame, matrix ou vector para .csv (dos 2 tipos csv e csv2) e para .txt.
A estrutura é semelhante para diferentes métodos (write.csv(), write.csv2() ou write.table()):
write.csv(Objeto, file = “objeto.csv", append = FALSE, quote = TRUE, sep = " ", eol = "\n",
na = "NA", dec = ".", row.names = TRUE, col.names = TRUE, qmethod = c("escape", "double"), fileEncoding = "")
Vamos experimentar os diferentes comandos. Antes de prosseguir certifique-se que o endereço de saída está adequado.
write.csv(Info.Alunos, file="J:/ENG 792/ENG_792-AVDR/ENG.792-AVDR/Cap_3_Informações - Alunos ENG 792.csv", sep=",",fileEncoding="UTF-8")## Warning in write.csv(Info.Alunos, file = "J:/ENG
## 792/ENG_792-AVDR/ENG.792-AVDR/Cap_3_Informações - Alunos ENG 792.csv", :
## attempt to set 'sep' ignored## Error in is.data.frame(x): objeto 'Info.Alunos' não encontradowrite.csv2(Info.Alunos, file="J:/ENG 792/ENG_792-AVDR/ENG.792-AVDR/Cap_3_Informações - Alunos ENG 792.2.csv",sep=",",fileEncoding="UTF-8")## Warning in write.csv2(Info.Alunos, file = "J:/ENG
## 792/ENG_792-AVDR/ENG.792-AVDR/Cap_3_Informações - Alunos ENG 792.2.csv", :
## attempt to set 'sep' ignored## Error in is.data.frame(x): objeto 'Info.Alunos' não encontradowrite.table(Info.Alunos, file="J:/ENG 792/ENG_792-AVDR/ENG.792-AVDR/Cap_3_Informações - Alunos ENG 792.txt", sep=",",fileEncoding="UTF-8")## Error in is.data.frame(x): objeto 'Info.Alunos' não encontrado4.3.8 Definir diretório de trabalho setwd()
Durante a importação e exportação de dados nós forncemos dentro dos comandos os endereço no seu computador ou periférico de onde e para onde os dados deveriam ser importados e exportados, respectivamente.
Esta tarefa pode ser simplificada pela determinação de um diretório de trabalho. Após esta definição o R vai comunicar diretamente com esta pasta de maneira que todos os arquivos importados e exportados de a para esta pasta.
setwd("J:/ENG 792/ENG_792-AVDR/ENG.792-AVDR/")
Al.Eng792<-read.xlsx(file="Cap_3_Informações - Alunos ENG 792.xlsx", endRow=28,sheetIndex=4,startRow = 2, header =T, encoding="UTF-8")
write.table(Reposição.de.aula, file="Cap_3_Informações - Alunos ENG 792.txt",sep=",",fileEncoding="UTF-8")
Explore o comandos rowSums(), colSums(), rowMeans(), colMeans() e acrescente no data.frame notas uma coluna com soma e média das notas dos alunos e linhas com no fim do arquivo soma e média das notas 1 a 4. Para terminar exporte os arquivos em formato .csv e .txt
Para mais informações sobre importação e exportação de dados leia RCoreTeam (2020) .
E digo mais, é só isso mesmo.
4.4 Exercícios
- Defina um objeto chamado
acom valores de 60 a 85. - Determine o elemento 12 de
a. - Determine elemento 20 de
a. - Mostre o 5° e 25° elementos de
a. - Mostre o 4°, 8°, 12°, 20° e 24° elementos de
a. - Mostre todos exceto 18°elemento de
a. - Mostre todos exceto o 2°, 15° e 17° elementos de
a. - Gere um data frame chamado
new_framecom 3 colunas: A primeira com valores de 33 a 47; a segunda começando em 115 e terminando em 157, com incrementos de 3; e a terceira começando em 10, com 15 elementods, e cada elemento decrescendo 8.5. Cada coluna deve ser chamada deX,YeZrespectivamente. - Extraia todos os valores da coluna 1.
- Extraia das linhas 6 a 10 da coluna 2.
- Extraia os valores da linha 11 para todas as colunas.
- Extraia os valores das linhas 4 a 8 das colunas 1 e 3 em formato de data frame
- Demonstre o número de linhas do
new_frame. - Demonstre 4 diferentes métodos de extrair os valores de todas as linhas da coluna 1 de
new_frame. - Crie uma função que multiplique os valores de cada coluna por 2 ao cubo e em seguida aplique para o módulo da mesma seleção
log10()criandonew_frame2alojando-o no global environment. - Exporte seu
new_frame2para .csv e .txt.
4.5 Dica de leitura
Introduction to the pacman Package in R (3 Examples)
Quick list of useful R packages
36 Instalando e usando pacotes (packages) do R
Installing and Using R Packages
15 Essential packages in R for Data Science
A Tutorial on Using Functions in R!
Writing Functions in R: Example One